Innholdsfortegnelse:
- Forfatter John Day day@howwhatproduce.com.
- Public 2024-01-30 11:23.
- Sist endret 2025-01-23 15:02.
Av: Madhumita Kannan, Henry Nguyen, Ashley Urrutia Avila, Mei Jin
Denne MATLAB -koden er et program for å oppdage den eksakte størrelsen, formen og plasseringen av en svulst som finnes i en pasients MR -skanninger i hjernen. Dette programmet er designet for opprinnelig å fungere med tumordeteksjon i hjernens MR -skanninger, men det kan også brukes til kreftdiagnostikk i andre organscanninger.
Følgende instruksjoner vil først beskrive metodene for bildeanalyse gjennom filtrering og opprydding av MR -skanning, gjennom binærisering, medianfiltrering og skyvevinduer. Deretter vil den instruere om hvordan man kan isolere svulsten ved hjelp av en forhåndsgenerert elliptisk maske, og filtrere den videre for å skissere omkretsen av svulstens form.
Når svulsten er oppdaget, vil instruksjonene ytterligere beskrive hvordan dette programmet skal inkorporeres i et grafisk brukergrensesnitt (GUI). Gjennom disse instruksjonene vil riktig kode og filer bli vedlagt for å forklare hvordan denne MR -skanneanalysen fungerer.
Noen ting å vite, laste ned og ha klar før du fortsetter med denne instruksen: 1. Sørg for å ha den nyeste versjonen av MATLAB lastet ned. Du kan installere R2018b her:
2. For å kjøre dette programmet må du ha tilgang til MR -hjerneskanningsfiler. Selv om noen alltid kan bli funnet fra Google -bilder, kan grundig og nøyaktig analyse utføres fra riktige bilder av forskjellige lag med hjerneskanninger for hver pasient. Du kan få tilgang til filene for 20 forskjellige pasienter med glioblastom før og etter behandling fra denne databasen:
3. Fokuset for dette programmet og de ulike metodene som veileder dette prosjektet er skissert i denne forskningsrapporten:
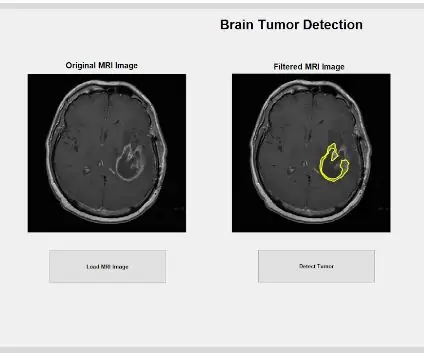
Trinn 1: Start grafisk brukergrensesnitt (GUI)
Det første trinnet vil være å lage og starte det grafiske brukergrensesnittet, GUI. Dette kan gjøres ved å skrive inn guide i kommandovinduet, trykke enter og opprette en ny GUI. Når dette trinnet er fullført, kan du begynne å lage funksjoner som akser, statisk tekst, redigere tekst og trykknapper som vises når programmet kjøres og brukeren kan samhandle med. Disse funksjonene kan redigeres og manipuleres gjennom eiendomsinspektøren, men den viktigste funksjonen som må endres når du oppretter disse funksjonene er Tag -navnet. Det er viktig å endre Tag -navnet til hver funksjon som er implementert, fordi det vil tillate oss å lage en gjenkjennelig tilbakeringingsfunksjon. Når du er fornøyd med utformingen av GUI -en, kan du gå videre til å laste inn DICOM -filene som skal vises i GUI.
Trinn 2: Laste og lese MR -bilder i MATLAB
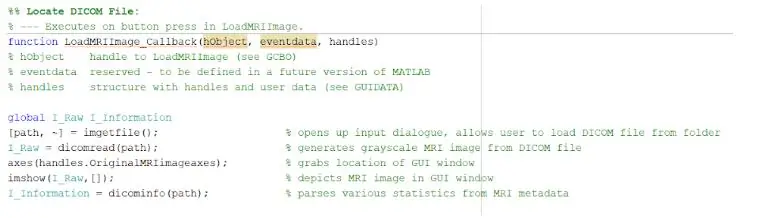
For å laste DICOM -filene må du initialisere tilbakeringingsfunksjonen som ville bli utført når du trykker på knappen "Last inn MRI -bilde." Når dette er fullført, må du opprette en global variabel som viser bildet på håndtakets akser der du vil at det originale MR -bildet skal vises. MR -skanningsbildene som lastes ned fra databasen er alle DICOM -formaterte filer som må lastes inn i MATLAB -katalogen. Finn filen ved hjelp av imgetfile for å laste dem inn i programmet. Bildene leses ved hjelp av den innebygde MATLAB -funksjonen 'dicomread', og det første råbildet for hver fil er innebygd i de venstre GUI -aksene ved hjelp av imshow.
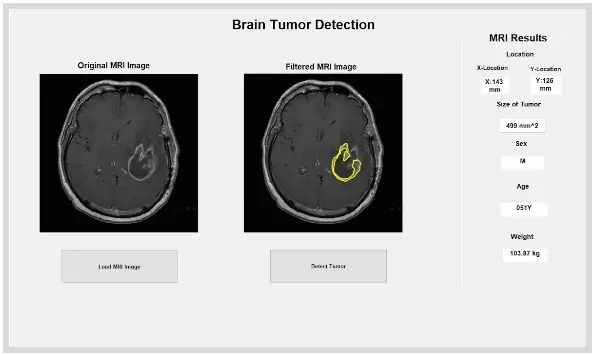
Den innebygde MATLAB -funksjonen 'dicominfo' er også ekstremt nyttig for å adressere all informasjonen for hver MR -dicom -fil. Vi brukte denne funksjonen til å trekke ut all den beskrivende informasjonen til pasientene, for eksempel kjønn, alder, vekt og høyde. Denne funksjonen gir deg også stabelordren som er nyttig for implementering av programmet i det grafiske brukergrensesnittet. Vi opprettet variabler for hver av de beskrivende opplysningene til pasientene som vil bli brukt for GUI når det trykkes på detekteringsknappen.
Trinn 3: Bildefiltrering

Når DICOM -filen til råbildet er lastet inn og lest, må bildet konverteres fra gråtoner til en binær form som består av bare svart -hvite piksler. Vi brukte funksjonen 'imbinarize' for å lage et binært bilde fra det rå bildet av kontrollere aspekter ved adaptiv terskling ved sensitivitetsverdi på 0,59. Standard terskelfølsomhetsfaktor, 0,5 var lav og klarte ikke å oppdage de lysere klattene og flekkene fra bildet, så vi økte den til 0,59.
Det binære bildet blir deretter behandlet gjennom et medianfilter ved hjelp av funksjonen 'medfilt2' fordi det binære bildet er todimensjonalt. Vi setter hver utgangspiksel til å inneholde medianverdien i 5 x 5 -nabolaget rundt den tilsvarende pikslen i det inngående binære bildet. Dette reduserer støyen og bevarer kantene i en 5 x 5 firkant rundt hver piksel. Deretter bruker vi et skyvevindu ved hjelp av 'strel', for å lage et skiveformet flatt strukturelement med en nabolagsradius på 2 for å identifisere hver sentral opprinnelsespiksel i hvert diskområde. Vi brukte et diskstruktureringselement fordi vi analyserer hvert sirkulære punkt og pikslene i hvert punkt, så et diskformelement er mer nyttig.
Når bildet er filtrert, kan det rengjøres ved hjelp av "lukk" -funksjonen for å fjerne de svarte flekkene mellom de filtrerte hvite pikslene i bildet, og lukker alle hullene rundt det. Det fullstendig bearbeidede bildet kan deretter plottes i den andre delplottet i den forhåndstildelte figuren, slik at en sammenligning kan gjøres mellom det rå og det filtrerte bildet.
Trinn 4: Tumorisolasjon gjennom elliptisk maske


Lyspunktene i svulsten kan deretter isoleres fra det hovedfiltrerte bildet gjennom en forhåndsgenerert elliptisk maske. For å lage denne masken, bør du kjenne størrelsen på det originale, rå MR-skanningsbildet, og ved å bruke rad- og kolonnelengden, som henholdsvis x og y- koordinater, tilordne senterkoordinatene for elliptikken. Vi setter y-aksen som en hovedakse med en radius på 50 enheter fra sentrum, og den mindre aksen med en radius på 40 enheter fra sentrum.
Vi brukte MATLAB-funksjonen 'meshgrid' til å generere et kartesisk plan med todimensjonale rutenettkoordinater basert på koordinatene i vektorer fra 1 til lengden på x-aksen, og fra 1 til lengden på y-aksen til bildet. Col er en matrise der hver rad er en kopi av x-aksen, og rad er en matrise der hver kolonne er en kopi av y-aksen. Det kartesiske rutenettet representert av koordinatene Col og Row har lengder (1: Y_Size) rader og lengde (1: X_Size) kolonner. Bruk indeksene til Col og Row generert av det kartesiske rutenettet for å bestemme ligningen for ellipse avhengig av den forhåndsbestemte radiusen og senterkoordinatene. Den elliptiske konturen kan nå fylles med de hvite pikslene som finnes fra svulstflekker.
Ved å bruke den forhåndsgenererte elliptiske masken kan vi beskjære den spesifikke svulsten du ønsker å analysere fra det filtrerte bildet. Den elliptiske masken oppdager hvilke flekker som logisk passer innenfor omrisset av ellipsen og godtar dette som et punkt på det filtrerte bildet for å være akseptabelt som en svulst. Funksjonen 'bwareafilt' filtrerer deretter ut alle andre objekter utenfor denne oppdagede svulsten fra bildet. Vi brukte et bestemt vindu på 500 x 4000 empirisk basert på dimensjonene til alle bildene. Vi brukte deretter et annet skyvevindu med "strel" som et flatt skiveformet strukturelement med en større nabolagradius på 6, for å lukke hullene mellom hver sentral hvit piksel i den detekterte svulsten. Det påviste svulstpunktet ryddes ytterligere opp ved å bruke ‘imclose’ for ytterligere å eliminere de svarte pikslene og fylle ut alle hullene med ‘imfill’. Denne behandlede svulsten kan deretter vises i den tredje delplottet i det forhåndsdelte plottet for å gi en sammenligning mellom den isolerte svulsten og de originale og filtrerte bildene av MR -skanningen.
Trinn 5: Tumor Outlining
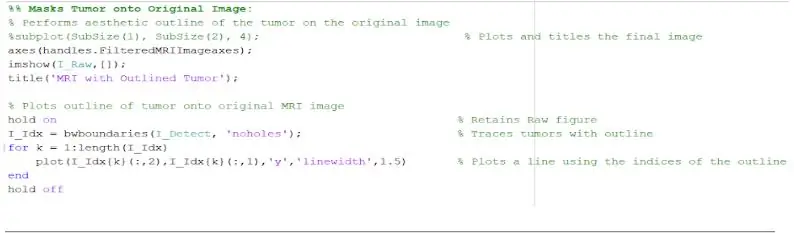
Nå som svulsten er isolert med masken, kan den skisseres og vises på det opprinnelige bildet, for å vise den nøyaktige plasseringen. For å gjøre dette brukte vi funksjonen 'bwboundaries' for å spore den tidligere oppdagede svulsten med en oversikt. Vi spesifiserte omrisset for ikke å inkludere hullene i tumorobjektet slik det blir skissert. Dette kan plottes på det originale, rå bildet, ved hjelp av en "for" -løkke som plotter omrisset rundt svulsten ved hjelp av indeksene på linjen med en linjebredde på 1,5 piksler. Denne konturen blir deretter plottet inn på det rå bildet, og viser den eksakte størrelsen og plasseringen av svulsten, i forhold til den opprinnelige MR -skanningen.
Trinn 6: Analysere de fysiske egenskapene til svulster
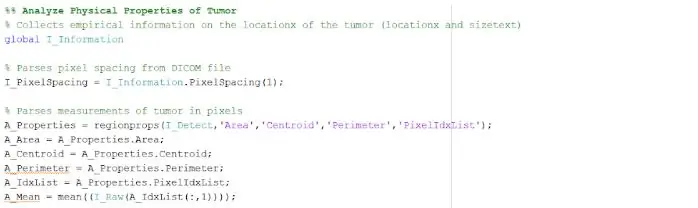
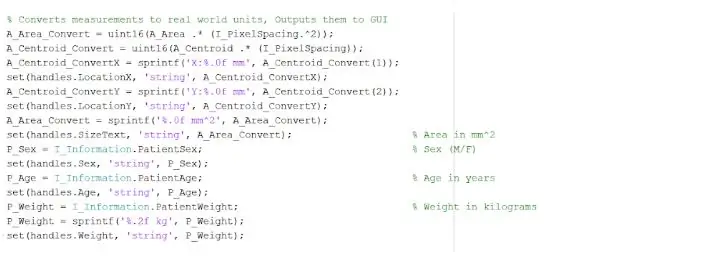
Det isolerte og skisserte stedet kan gi oss nyttig informasjon om svulstens størrelse, område og plassering. Vi brukte funksjonen ‘regionprops’ til å oppdage egenskapene til svulsten som angår areal, omkrets, sentroider og pikselindeksverdien. Denne pikselindeksverdien gir oss den virkelige verdenenheter for hver piksel i hvert bilde, unik for hver skanning. Disse egenskapene kan deretter konverteres til virkelige enheter på millimeter. Den empiriske informasjonen programmet gir oss er unik for hver MR -skanning og er ekstremt nyttig for å bestemme størrelsen, plasseringen og typen svulst som brukerne kan analysere og innlemme i det grafiske brukergrensesnittet.
Anbefalt:
DIY -- Hvordan lage en edderkopprobot som kan kontrolleres ved hjelp av smarttelefon ved hjelp av Arduino Uno: 6 trinn

DIY || Hvordan lage en edderkopprobot som kan kontrolleres ved hjelp av smarttelefon ved hjelp av Arduino Uno: Mens du lager en edderkopprobot, kan du lære så mange ting om robotikk. Som å lage roboter er både underholdende og utfordrende. I denne videoen skal vi vise deg hvordan du lager en Spider -robot, som vi kan bruke ved hjelp av smarttelefonen vår (Androi
Få blind til å gjenkjenne ting ved å berøre ting rundt dem ved hjelp av MakeyMakey: 3 trinn

Gjør blinde gjenkjenne ting ved å berøre ting rundt dem ved hjelp av MakeyMakey: introduksjon Dette prosjektet tar sikte på å gjøre blindes liv lett ved å identifisere ting rundt dem gjennom berøringsfølelsen. Meg og min sønn Mustafa tenkte vi på å finne et verktøy for å hjelpe dem, og i perioden vi brukte MakeyMakey -maskinvaren til
Kontrollere Raspberry Pi ved hjelp av Matlab: 5 trinn

Kontrollere Raspberry Pi ved hjelp av Matlab: Hei, denne opplæringen handler om å kontrollere din hodeløse bringebær pi ved hjelp av matlab. Du må kanskje installere matlab til den nyeste versjonen for støtte til nyere bringebær pi -bord
Hvordan lage en drone ved hjelp av Arduino UNO - Lag en quadcopter ved hjelp av mikrokontroller: 8 trinn (med bilder)

Hvordan lage en drone ved hjelp av Arduino UNO | Lag en Quadcopter ved hjelp av mikrokontroller: Introduksjon Besøk min Youtube -kanal En Drone er en veldig dyr gadget (produkt) å kjøpe. I dette innlegget skal jeg diskutere hvordan jeg får det billig? Og hvordan kan du lage din egen slik til en billig pris … Vel, i India er alle materialer (motorer, ESCer
Hvordan lage en hørselstest for voksne ved hjelp av MATLAB: 6 trinn

Hvordan lage en hørselstest for voksne ved bruk av MATLAB: ANSVARSFRASKRIVELSE: Testen vår er IKKE en medisinsk diagnostikk og bør ikke brukes som sådan. For å måle hørselen nøyaktig, vennligst kontakt en lege. Ved hjelp av materialer vi allerede hadde, foretok gruppen vår en hørselstest. Testen vår er kun til bruk for voksne og tenåringer
