Innholdsfortegnelse:
- Trinn 1: Organisering av mammogramdata
- Trinn 2: Bildebehandling
- Trinn 3: Terskelkode
- Trinn 4: Finne abnormiteter for hvert binært bilde
- Trinn 5: Plotte den diagnostiserte masseposisjonen og størrelsen for visuell sammenligning
- Trinn 6: Implementering av den andre sammenligningsmetoden
- Trinn 7: Analyse av innsamlede data
- Trinn 8: Lag din egen klassifisering
- Trinn 9: Forbedringer? Noen tanker?
- Forfatter John Day day@howwhatproduce.com.
- Public 2024-01-30 11:23.
- Sist endret 2025-01-23 15:02.
Formålet med dette prosjektet var å identifisere og bruke en parameter for å behandle gråtonemammogrammer av forskjellige bakgrunnsvevsklassifiseringer: Fatty, Fatty Glandular, & Dense Tissue. Denne klassifiseringen brukes når radiologer analyserer mammogrammer og må vurdere om vevstetthet vil skjule eventuelle abnormiteter som lesjoner eller svulster. Dette er fordi både normale fysiologiske strukturer som kjertelvev og fibrøst bindevev. og unormale morfologier som forkalkninger og svulster vil se veldig lyse ut på mammogrammet, mens mindre tett fettvev vil se svart ut. Derfor var det hensiktsmessig å programmere en klassifikator som kan manipulere pikselintensitetsnivåer for best å visualisere og identifisere masser.
Trinn 1: Organisering av mammogramdata
En av de første tingene jeg innså at jeg trengte å håndtere var å organisere dataene på en veldig tydelig, kortfattet og tilgjengelig måte. Dette er variablene jeg hentet fra mini-MIAS-databasen for mammogrammer. Jeg opprettet to matriser. En som inneholder 4 kolonner:
- Bilde nummer:
- x massekoordinat
- y massekoordinat
- Masseradius: (Dette definerte en omtrentlig størrelse for massen
Den andre serien inneholdt klassifiseringsinformasjon:
- Type bakgrunnsvev: Fett (F), Fettkirtel (G), Tett (D)
- Beskrivelse av masse: Veldefinert (CIRC), spikulert (SPIC), dårlig definert annen (MISC) Arkitektonisk forvrengning (ARCH), Asymmetri (ASYM), Normal (NORM)
- Diagnoser: Godartet (B), ondartet (M)
Siden målet med dette prosjektet var å bestemme den beste terskelen for hver type bakgrunnsvev, var ikke all informasjon nødvendig. Imidlertid kan du utvide prosjektet ditt til å inkludere teksturanalyse og teste klassifikatoren mot de kjente massebeskrivelsene.
Sidebemerkning: Databasen som jeg fikk diagnostiserte mammogrambilder fra, organiserte informasjonen om hvert mammogram i en tekstfil atskilt fra bildene. Det var mildt sagt vanskelig for meg å trekke ut dataene fra en tekstfil og organisere i matriseskjemaer, men følgende lenke var veldig nyttig for å finne ut av alt det. Alternativt kan du bare justere koden jeg limte inn ovenfor for dine formål.
Mammogramfilformat: mdb001 G CIRC B 535 425 197
mdb002 G CIRC B 522 280 69
TextScan Help: https://www.mathworks.com/help/matlab/ref/textsca…Mammogram Database:
Trinn 2: Bildebehandling
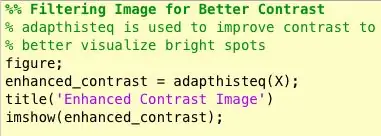
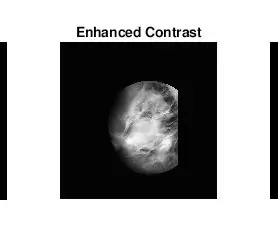
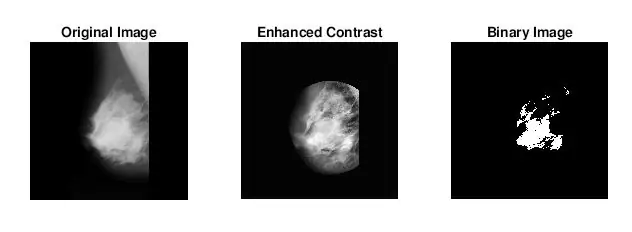
Den andre tingen som dukket opp da jeg fant ut hvordan jeg identifiserte masser var at jeg for mange unormale mammogrammer ikke visuelt kunne se hvor abnormiteten var eller hvor stor den var. Selvfølgelig, ettersom jeg ikke er en erfaren radiolog, var det forventet. Imidlertid var den mest enkle måten å finne abnormiteter (i henhold til mine lange google -søk) å se på konsentrasjoner av lyse og mørke områder. Jeg brukte først og fremst adapthisteq -funksjonen for å forbedre bildekontrasten og deretter imbinarisere for å konvertere bildet til et binært bilde for å eksperimentere med forskjellige terskelnivåer.
- adapthisteq: Denne funksjonen transformerer intensitetsverdier for gråtoner og rgb -bilder ved hjelp av kontrastbegrenset adaptiv histogramutjevning. Med andre ord, det justerer histogrammet for intensitetsverdier til en spesifisert distribusjonstype. Matematikklinken for denne funksjonen er vedlagt nedenfor for videre lesing.
- imbinarize: lager et binært bilde fra et gråtonebilde ved å tilordne alle piksler over en viss intenisti til 1s og pikslene under den verdien a 0. Jeg brukte denne funksjonen til å teste den optimale terskelen for å redusere bakgrunnsvevstøy.
Trinn 3: Terskelkode
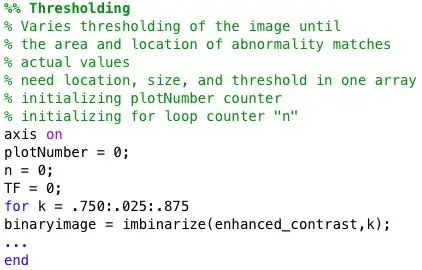
En for sløyfe brukes til å binærisere mammogrammet med varierende terskler. For å gi et større bilde, inneholder for -løkken koden fra trinn 3 til trinn 7. Så hvert binære bilde vil bli analysert for abnormiteter. I tillegg er dette for loop innkapslet i et annet for loop som importerer et nytt mammogrambilde fra databasen i hver iterasjon.
Trinn 4: Finne abnormiteter for hvert binært bilde
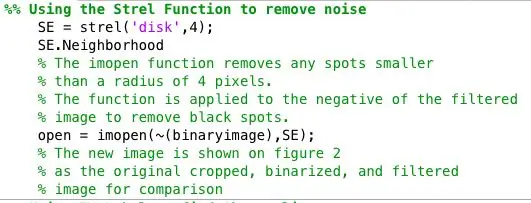
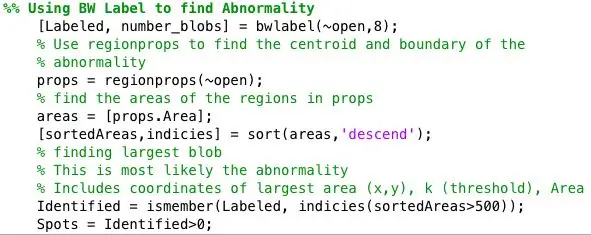
Jeg behandlet de binære bildene videre ved hjelp av strel -funksjonen i forbindelse med imopen for å fjerne bakgrunnsstøy. Det binære bildet fra forrige trinn inverteres og filtreres ved hjelp av nabolaget definert av SE. Deretter brukte jeg bwlabel til å merke alle områder som hadde minst 8 tilkoblede piksler.
Regionstøttefunksjonen ble brukt til å finne sentroidene og egenskapene til hvert sted identifisert av bwlabel.
Da ble alle flekker større enn 500 piksler identifisert ved hjelp av ismember. Sentroidene for de identifiserte flekkene ble plottet på et bilde som bare viste flekkene større i areal enn 500. Area Identified = ismember (Labeled, indicies (sortedAreas> 500)); Flekker = identifisert> 0;
Trinn 5: Plotte den diagnostiserte masseposisjonen og størrelsen for visuell sammenligning
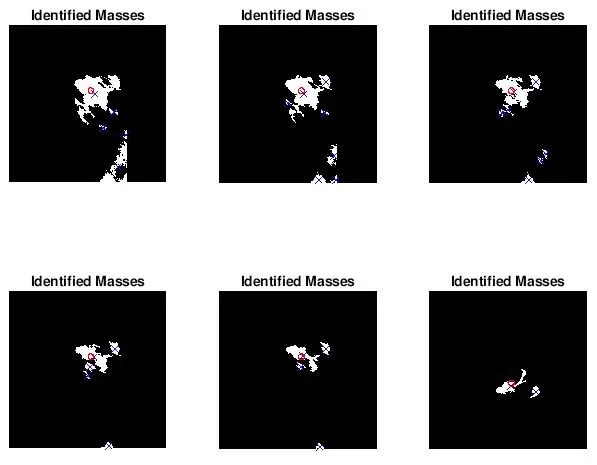
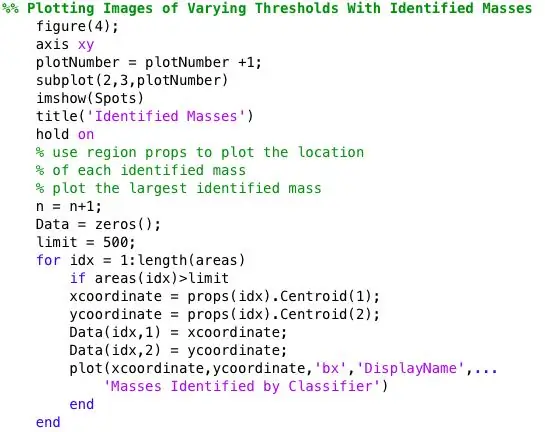

Jeg ønsket å se om flekkene funnet av bwlabel var riktige. Jeg gjorde dette på to måter. Jeg analyserte først nøyaktigheten til klassifisereren min ved å gjøre en visuell sammenligning. Jeg tegnet ganske enkelt den faktiske størrelsen og plasseringen av abnormiteten (rød sirkel) og plasseringen bestemt av koden (blå x) på det forhåndsbehandlede mammogrambildet. De seks bildene ovenfor viser effekten av å øke grenseverdien.
Trinn 6: Implementering av den andre sammenligningsmetoden

Den andre måten jeg testet klassifisereren og terskelverdiene på var ved å bestemme om stedene som ble funnet av klassifisereren var innenfor en viss avstand fra de diagnostiserte abnormitetskoordinatene. Jeg lagret tersklene som minst ett av de identifiserte punktene var innenfor 1,5*r fra den kjente abnormiteten til en egen tekstfil som heter Mammogram Data. Hensikten med dette var å finne minimumsterskelen som trengs for at klassifisereren min kan identifisere abnormiteten.
Trinn 7: Analyse av innsamlede data
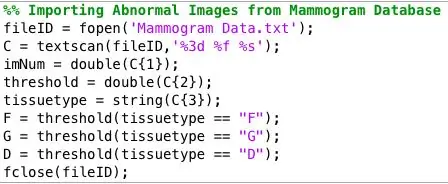
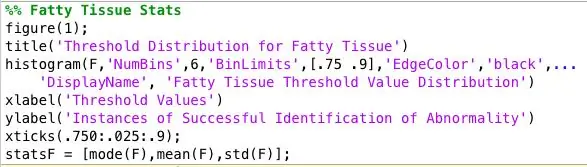
Jeg kjørte programmet på alle de unormale mammogrambildene, og jeg satt igjen med en enorm tekstfil med data. For å finne den beste terskelen for hver type vev, organiserte jeg dataene etter vevstype og tegnet et histogram over terskelverdiene for hver vevstype. Den riktige terskelverdien ble bestemt på hvilken terskel som ga de mest nøyaktige resultatene for hver vevstype. Jeg lagret disse dataene for å laste opp til klassifisereren min.
Trinn 8: Lag din egen klassifisering

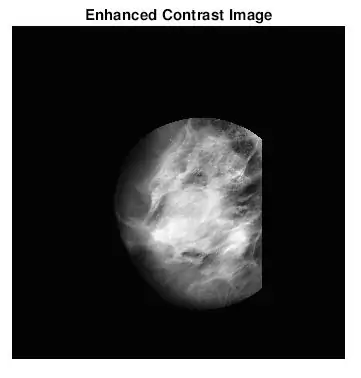
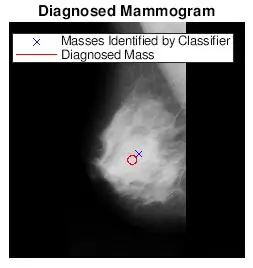
Etter at jeg fant de mest passende terskelverdiene for hver vevstype, redigerte jeg min originalkode for å få en bruker til å angi bildetall og vevstype for å velge terskelen for mammogrambildet. Jeg plottet deretter det diagnostiserte mammogramstedet med de funnet stedene på de originale mammogrambildene. Jeg ønsket å gjøre dette morsommere, så jeg programmerte en funksjon for å beskjære et sirkulært område rundt ROI. Brukeren vil bli bedt om å velge et midtpunkt og flere punkter som best omfatter ROI. Jeg har lagt ved begge matlab -filene her.
Trinn 9: Forbedringer? Noen tanker?
Da jeg skrev dette instruerbare, begynte jeg å se mange forbedringer jeg kunne gjøre i klassifiseringen, for eksempel å finne måter å skille mellom forskjellige typer masser identifisert basert på teksturanalyse eller forbedre testingen min for nøyaktighetsdel av SandBoxProject. fil. Siden dette var et prosjekt med en frist, måtte jeg stoppe et sted, men jeg håper at jeg kan bruke bildebehandlingsferdighetene jeg lærte i andre applikasjoner. Jeg har også vedlagt filen som ble brukt til å batchbehandle alle de unormale mammogrambildene.
Anbefalt:
Hvordan bruke DS1307 Bruke Arduino: 7 trinn

Hvordan bruke DS1307 Bruke Arduino: DS1307 er en sanntidsklokke IC (RTC). Denne IC brukes til å gi tidsdata. Tildelt tid starter fra sekunder, minutt, timer, dager, dato, måned og år. Denne IC krever ytterligere eksterne komponenter som Crystal og 3,6V batterier. Krystall
Visualisere barometrisk trykk og temperatur ved hjelp av Infineon XMC4700 RelaxKit, Infineon DPS422 og AWS .: 8 trinn
Visualisere barometertrykk og temperatur ved bruk av Infineon XMC4700 RelaxKit, Infineon DPS422 og AWS .: Det er et enkelt prosjekt for å fange barometrisk trykk og temperatur ved hjelp av Infineon's DPS 422. Det blir klønete å spore trykk og temperatur over en periode. Det er her analytics kommer inn i bildet, innsikt i endringen i
Hvordan bruke en tenåring til å skrive ut bilder på Splatoon 2 Bruke SplatPost -skriveren: 10 trinn

Hvordan bruke en tenåring til å skrive ut bilder på Splatoon 2 Bruke SplatPost -skriveren: I denne instruksen vil jeg vise hvordan du bruker SplatPost -skriveren av ShinyQuagsire. Uten klare instruksjoner ville noen som ikke har erfaring med kommandolinjen ha litt problemer. Målet mitt er å forenkle trinnene ned til poi
Lese ultralydsensor (HC-SR04) Data på en 128 × 128 LCD og visualisere den ved hjelp av Matplotlib: 8 trinn
Lese ultralydsensor (HC-SR04) Data på en 128 × 128 LCD og visualisere den ved hjelp av Matplotlib: I denne instruksen vil vi bruke MSP432 LaunchPad + BoosterPack til å vise en ultralydsensors (HC-SR04) data på en 128 × 128 LCD og send dataene til PC serielt og visualiser dem ved hjelp av Matplotlib
Neopixel Ws2812 Rainbow LED-glød med M5stick-C - Kjører Rainbow på Neopixel Ws2812 Bruke M5stack M5stick C Bruke Arduino IDE: 5 trinn

Neopixel Ws2812 Rainbow LED-glød med M5stick-C | Running Rainbow på Neopixel Ws2812 Bruke M5stack M5stick C Bruke Arduino IDE: Hei folkens i denne instruksen, vi lærer hvordan du bruker neopixel ws2812 LED eller led strip eller led matrise eller led ring med m5stack m5stick-C utviklingstavle med Arduino IDE, og vi skal lage et regnbuemønster med den
