Innholdsfortegnelse:
- Forfatter John Day day@howwhatproduce.com.
- Public 2024-01-30 11:21.
- Sist endret 2025-01-23 15:02.
Av: Phuc Lam, Paul Yeung, Eric Reyes
Erkjenner at feil i lungesegmentering vil gi falsk informasjon om identifisering av et sykdomsområde og kan direkte påvirke diagnoseprosessen. Moderne datamaskinteknikker ga ikke nøyaktige resultater når lungesykdommer har utfordrende former. Disse unormale formene kan være forårsaket av pleural effusjoner, konsolideringer etc. Ved å bruke lungesegmenteringsteknikken, der lungens grenser er isolert fra omkringliggende thoraxvev, kan appen vår identifisere grensene med brukerens inngangsterskler for å gi fullt tilpassbare visninger av formene på lungene, Hensikten med dette MatLab-prosjektet er å lage en brukervennlig interaktiv lungesegmenteringsapp for å oppdage patologiske forhold i lungers røntgenbilder. Målet vårt er å skape en mer effektiv måte å illustrere og identifisere unormale lunger for å gi leger og radiologer en mer pålitelig måte å diagnostisere lungesykdommer. Ved å bruke appdesignerverktøyet i MatLab, er programmet designet for å fungere spesielt med røntgenbilder av brystet og computertomografi (CT), men det er også testet for å fungere med MR-skanninger.
Instruksjonene nedenfor inneholder vår støyfiltreringsteknikk (lavpass Wiener-filter) samt bildeterskelen (ved å bruke intensitetshistogrammet til gråtonebildet) og ved å bruke en morfologisk gradient (forskjellen mellom utvidelse og erosjon av et bilde) til identifisere en region av interesse. Instruksjonen vil deretter forklare hvordan vi integrerer alle elementene i det grafiske brukergrensesnittet (GUI).
Merk:
1). Dette prosjektet er inspirert av en forskningsartikkel: "Segmentering og bildeanalyse av unormale lunger ved CT: nåværende tilnærminger, utfordringer og fremtidige trender". Som du finner her
2). Vi bruker røntgenbilder fra NIH: Clinical Center. Link finner du her
3). Hjelp til appdesignere finner du her
4). Før du kjører koden: du må endre Dir -banen (på linje 34) til filkatalogen og type bilde (linje 35) (vi analyserer *.png).
Trinn 1: Trinn 1: Laster inn bilde
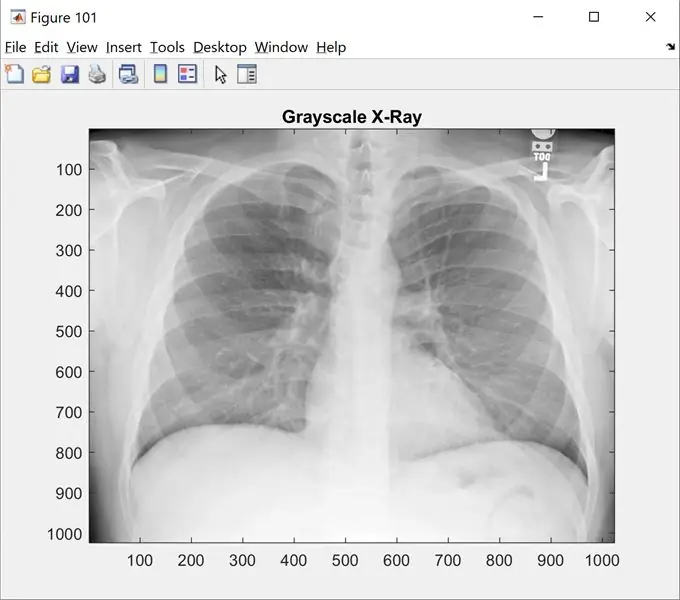
Dette trinnet viser deg det opprinnelige bildet i gråskala. Endre 'name_of_picture.png' til bildetavnet ditt
klar; clc; Lukk alle;
%% Laster inn bilder
raw_x_ray = 'name_of_picture.png';
I = imread (raw_x_ray);
figur (101);
imshow (I);
fargekart (grå);
tittel ('gråtonerøntgen');
Trinn 2: Trinn 2: Støyfiltrering og histogram
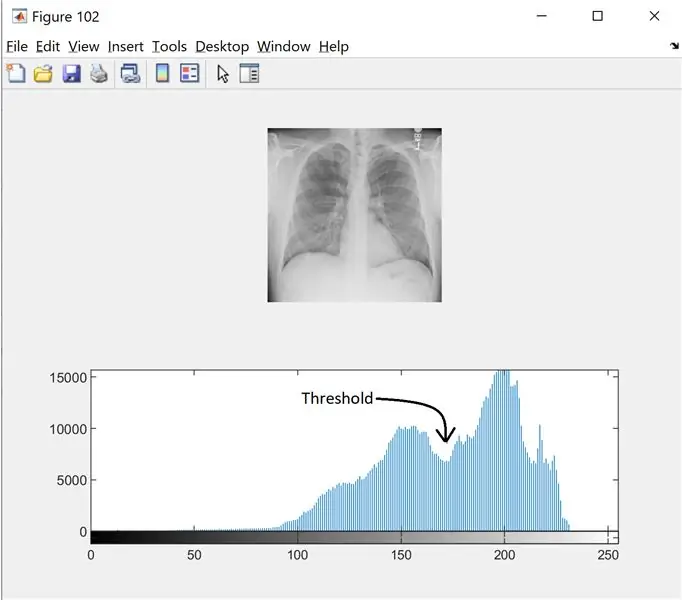
For å finne terskelen for gråskala -bildet, ser vi på histogrammet for å se om det er forskjellige moduser. Les mer her
I = wiener2 (I, [5 5]);
figur (102);
delplot (2, 1, 1);
imshow (I);
delplot (2, 1, 2);
imhist (I, 256);
Trinn 3: Trinn 3: Angi terskler
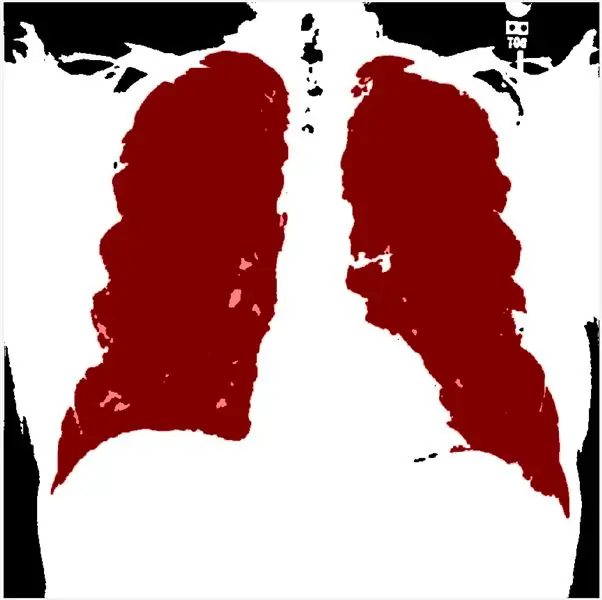
Dette trinnet lar deg sette terskelen i henhold til histogrammet. morfologiskGradient vil markere regionen av interesse for rødt, og funksjonen grensene overlapper det skisserte og filtrerte bildet av lungen i rødt.
Ved å bruke regionprops kan vi presisere soliditetens matriser og sortere dem synkende. Deretter binariserer jeg det grå sclae -bildet og bruker den morflogiske gradientmetoden og mLoren Shurasking for å markere interesseområdet (ROI). Det neste trinnet er å snu bildet slik at lunge -avkastningen er hvit i svart bakgrunn. Jeg bruker funksjonen showMaskAsOverlay for å vise 2 masker. Merk: koden er inspirert av Loren Shure, lenke.
Lasly, jeg lager rød kontur ved å bruke bwbwboundaries og maskere filterbildet og grensene.
a_thresh = I> = 172; % angir denne terskelen
[labelImage, numberOfBlobs] = bwlabel (a_thresh);
rekvisitter = regionprops (a_thresh, 'all');
sortedSolidity = sort ([props. Solidity], 'descend');
SB = sortedSolidity (1);
hvis SB == 1 % SB bare godtar soliditet == 1 filtrer ut bein
binaryImage = imbinarize (I); figur (103);
imshow (binaryImage); fargekart (grå);
SE = strel ('square', 3);
morphologicalGradient = imsubtract (imdilate (binaryImage, SE), imerode (binaryImage, SE));
mask = imbinarize (morfologisk gradient, 0,03);
SE = strel ('square', 2);
mask = imclose (mask, SE);
maske = fyll ut (maske, 'hull');
mask = bwareafilt (mask, 2); % kontroll antall områdeshow
notMask = ~ maske;
maske = maske | bwpropfilt (notMask, 'Area', [-Inf, 5000 - eps (5000)]);
showMaskAsOverlay (0,5, maske, 'r'); % du må laste ned app/funksjon showMaskAsOverlay
BW2 = imfill (binaryImage, 'hull');
nytt_bilde = BW2;
nytt_bilde (~ maske) = 0; % inverter bakgrunn og hull
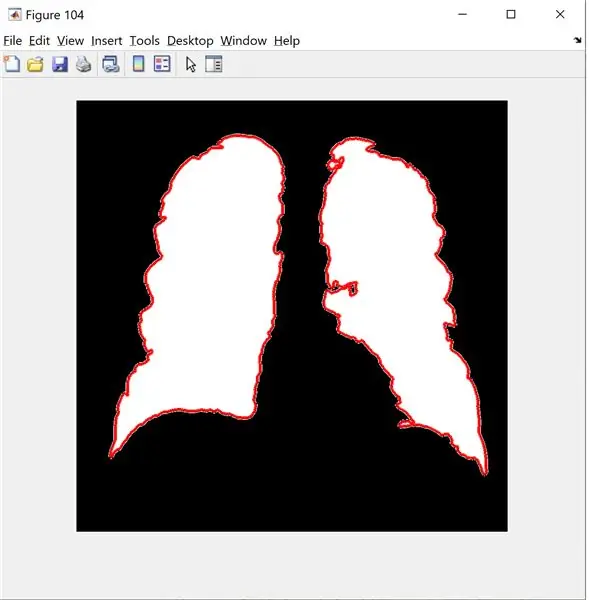
B = bwboundaries (new_image); % kan bare godta 2 dimensjoner
figur (104);
imshow (nytt_bilde);
vent litt
grenser (B);
slutt
Trinn 4: Opprette GUI
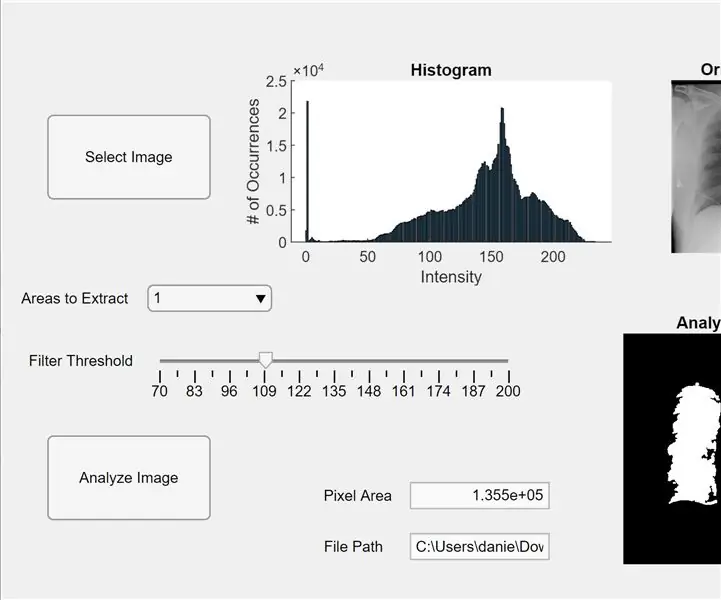
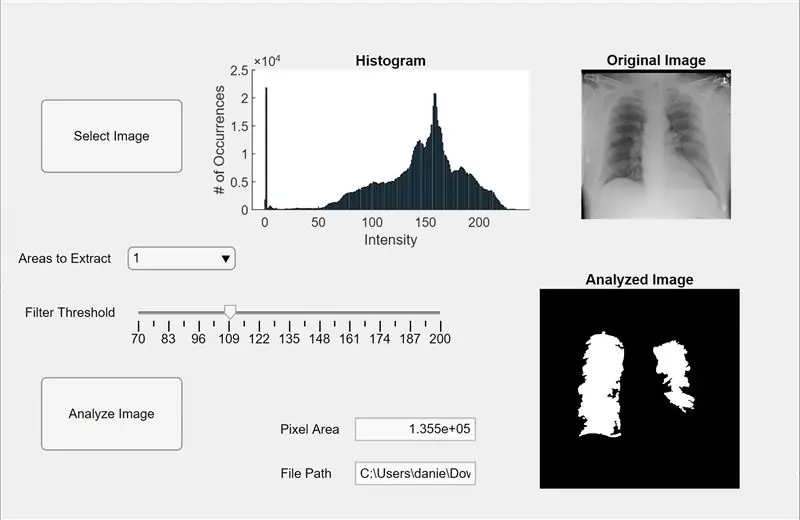
Nå integrerer vi den tidligere koden i en MATLAB -app. Åpne App Designer i MATLAB (Ny> App). Først designer vi grensesnittet ved å klikke-hold-og dra i tre akser inn i det midtre arbeidsområdet. Deretter klikker vi på og holder to knapper, ett redigeringsfelt (tekst), ett redigeringsfelt (numerisk), en glidebryter og en rullegardinmeny. To akser vil hver vise forhåndsvisningen og analysere bildet, og den tredje aksen vil vise et histogram med piksler for forhåndsvisningen "valgt" bilde. Redigeringsfeltet (tekst) -boksen viser filbanen til det valgte bildet, og redigeringsfeltet (numerisk) viser det oppdagede pikselområdet til lungene.
Bytt nå fra designvisning til kodevisning i App Designer. Skriv inn koden for eiendommer ved å klikke på den røde "Egenskaper" -knappen med et plusstegn ved den. Initialiser egenskapene I, terskel og regionsToExtract som i koden nedenfor. Høyreklikk deretter på en knapp øverst til høyre i arbeidsområdet (komponentleseren) og gå fra Tilbakeringinger> Gå til … tilbakeringing. Legg til koden for “function SelectImageButtonPushed (app, hendelse).” Denne koden lar deg velge et bilde som skal analyseres fra datamaskinen din ved hjelp av uigetfile. Etter at du har valgt et bilde, vises et forhåndsvisningsbilde under aksene ledsaget av et histogram. Høyreklikk deretter på den andre knappen og gjenta samme prosedyre for å opprette en tilbakeringingsfunksjon.
Legg til koden under “function AnalyzeImageButtonPushed (app, event).” Denne koden vil utføre pixeltelling og blobdeteksjon på forhåndsvisningsbildet på analysebildeknappen (den du høyreklikker for denne koden). Etter programmering av knappene, vil vi nå programmere glidebryteren og rullegardinmenyen. Høyreklikk på glidebryteren, opprett en tilbakeringingsfunksjon og legg inn koden under “function FilterThresholdSliderValueChanged (app, event)” til slutten. Dette gjør at glidebryteren kan justere terskelen for grå intensitet.
Lag en tilbakeringingsfunksjon for rullegardinmenyen, og legg til koden under “function AreastoExtractDropDownValueChanged (app, event)” for å la rullegardinmenyen endre antall blobber som vises på de analyserte bildeaksene. Klikk nå på hver enhet i komponentleseren og endre egenskapene til din smak, for eksempel å endre navn på enhetene, fjerne akser og endre skalering. Dra og slipp enhetene i komponentleseren i Design View til et funksjonelt og lettfattelig oppsett. Du har nå en app i MATLAB som kan analysere bilder av lunger for pikselområde!
eiendommer (Tilgang = privat) I = ; % bildefil
terskel = 257; %terskel for binærisering av grå intensitet
regionToExtract = 2;
slutt
funksjon SelectImageButtonPushed (app, hendelse)
clc; Dir = 'C: / Users / danie / Downloads / images_004 / images'; %definere uforandret fil "prefiks"
[imageExt, path] = uigetfile ('*. png'); %ta tak i den variable delen av bildetavnet
imageName = [Dir filesep imageExt]; %sammenkoblede variasjoner og variabler
app. I = imread (imageName); %leste bildet
imshow (app. I, 'parent', app. UIAxes); %viser bildet
app. FilePathEditField. Value = bane; %visningsfilbane for det opprinnelige bildet fra
slutt
function AnalyzeImageButtonPushed (app, hendelse)
originalImage = app. I;
originalImage = wiener2 (app. I, [5 5]); %prikkfjerningsfilter
histogram (app. AxesHistogram, app. I, 256); %visningshistogram for bildet
a_thresh = originalImage> = app.threshold; % angir denne terskelen
labelImage = bwlabel (a_thresh);
rekvisitter = regionprops (a_thresh, 'all');
sortedSolidity = sort ([props. Solidity], 'descend');
SB = sortedSolidity (1);
hvis SB == 1 % SB bare godtar soliditet == 1 filtrer ut bein
SE = strel ('square', 3);
morphologicalGradient = imsubtract (imdilate (labelImage, SE), imerode (labelImage, SE));
mask = imbinarize (morfologisk gradient, 0,03);
SE = strel ('square', 2);
mask = imclose (mask, SE);
maske = fyll ut (maske, 'hull');
mask = bwareafilt (mask, app.regionsToExtract);
% kontroll antall områdeshow
notMask = ~ maske;
maske = maske | bwpropfilt (notMask, 'Area', [-Inf, 5000 - eps (5000)]);
BW2 = imfill (labelImage, 'hull');
nytt_bilde = BW2;
nytt_bilde (~ maske) = 0;
B = bwgrenser (nytt_bilde); % kan bare godta 2 dimensjoner imshow (new_image, 'parent', app. UIAxes2);
hold (app. UIAxes2, 'on');
grenser (B);
sett (gca, 'YDir', 'reverse');
lungArea = bwarea (new_image);
app. PixelAreaEditField. Value = lungArea;
slutt
slutt
funksjon FilterThresholdSliderValueChanged (app, hendelse)
app.threshold = app. FilterThresholdSlider. Value;
slutt
function AreastoExtractDropDownValueChanged (app, event) stringNumber = app. AreastoExtractDropDown. Value;
app.regionsToExtract = str2double (stringNumber);
slutt
slutt
Anbefalt:
MATLAB Enkel ansiktsgjenkjenning: 4 trinn

MATLAB Enkel ansiktsgjenkjenning: Hovedmålet med disse instruktørene er å vise hvor lett bildebehandlingen vil være. Ved hjelp av MATLABFace har deteksjon og sporing vært et viktig og aktivt forskningsfelt, så derfor skal jeg forklare hvordan kan det gjøres med
Hvordan kode og publisere Matlab 2016b til Word (nybegynnerguide): 4 trinn

Hvordan kode og publisere Matlab 2016b til Word (nybegynnerguide): Matlab er et språk med høy ytelse som brukes til å beregne tekniske resultater. Den har muligheten til å integrere grafikk, beregninger og programmering på en brukervennlig måte. Med dette programmet kan brukeren publisere problemer og løsninger
Kontrollere Raspberry Pi ved hjelp av Matlab: 5 trinn

Kontrollere Raspberry Pi ved hjelp av Matlab: Hei, denne opplæringen handler om å kontrollere din hodeløse bringebær pi ved hjelp av matlab. Du må kanskje installere matlab til den nyeste versjonen for støtte til nyere bringebær pi -bord
Matlab-basert ROS Robotic Controller: 9 trinn
Matlab-basert ROS Robotic Controller: Helt siden jeg var barn har jeg alltid drømt om å være Iron Man og gjør det fortsatt. Iron Man er en av de karakterene som er realistisk mulig og rett og slett ønsker jeg å bli Iron Man en dag, selv om folk ler av meg eller sier det er umulig
Treningsprogrammeringsforbedringer i MATLAB: 6 trinn

Treningsprogrammeringsforbedringer i MATLAB: Mål: Målet med dette programmerte systemet er å se på en liten skala Arduino og bruke kodingen i større skala for å potensielt forbedre visse sikkerhetsfunksjoner i Amtrak Railroad -systemer. For å gjøre dette har vi lagt til en jordfuktighet sen
