Innholdsfortegnelse:
- Trinn 1: Bruke Brain Box
- Trinn 2: Intro: Analysere tverrsnitt
- Trinn 3: Konfigurere en funksjon: LevelCurveTracings.m
- Trinn 4: Utvikle den bundne algoritmen: Finne grenser
- Trinn 5: Utvikle den bundne algoritmen: Generere ytre bundet matrise
- Trinn 6: Utvikle den bundne algoritmen: Arbeide med senterpunkt
- Trinn 7: Utvikle den bundne algoritmen: Når en sentroid ikke er sentrert
- Trinn 8: Utvikle den bundne algoritmen: Interferens av hull
- Trinn 9: Utvikle den bundne algoritmen: Finne hull, fullføre hjernegrenser og hullgrenser
- Trinn 10: Loggdata: Funksjon PatientFiles.m
- Trinn 11: Logge data i filer
- Trinn 12: Loggdata: Visning av en plott av hjernevolum over tid
- Trinn 13: Lukking av hull i delplott: Subplotclose.m
- Trinn 14: Hovedkoden: Fjern alle og be om innspill
- Trinn 15: Hovedkoden: Batchbehandling av bildene
- Trinn 16: Hovedkoden: Polstring
- Trinn 17: Hovedkoden: Bestemmelse av grenser
- Trinn 18: Hovedkoden: Bestemmelse av riktig Z -dimensjon
- Trinn 19: Hovedkoden: Bestemmelse av X- og Y -koordinater
- Trinn 20: Hovedkoden: Plotte en tredimensjonal struktur, finne volum og loggdata
- Forfatter John Day day@howwhatproduce.com.
- Public 2024-01-31 10:23.
- Sist endret 2025-01-23 15:02.
Fremskrittet til grensen til lengre menneskeliv har ført til fremveksten av sykdommer som ikke er sett av sivilisasjoner før vårt. Blant disse påvirket Alzheimer omtrent 5,3 millioner levende eldre amerikanere i 2017, eller omtrent 1 av 10 eldre amerikanere (https://www.alz.org/facts/) og utallige andre med demens. For å hjelpe i kampen for å forstå hva som rammer våre eldste, vil denne koden utstyre fremtidige forskere og ivrige nysgjerrige med evnen til å spore hjernevolum over tid.
Trinn 1: Bruke Brain Box
For å bruke hjerneboks trenger man bare følgende:
- MR -skanning av en hjerne og navn og format på slike filer (skal alle ha omtrent de samme dimensjonene)
- Lengde på en skanning
- Avstand mellom hvert lag (MR -skanning)
- Pasientnavn (Ikke legg inn mellomrom ved innlegging, og bruk store og små bokstaver fornavn og etternavn, for eksempel: Fornavn Etternavn)
Og fra dette har man muligheten til å holde oversikt over individers trender i hjernevolum over tid. Dermed kan tall for Alzheimers trender spores av denne programvaren. Lengden vi brukte i forsøket var 180 mm for lengden på en skanning og 5 mm for avstanden mellom MR -skanninger, basert på gjennomsnittlige tall.
Imidlertid trenger applikasjonen av hjerneboks ikke å være begrenset til denne ene oppgaven. Hvis tverrsnittene av et gitt fast stoff er fotografier, som en svulst i seg selv, kan trendene i volumendringer for disse også spores i programvaren.
Trinn 2: Intro: Analysere tverrsnitt
I tredimensjonale strukturer vil de todimensjonale planene som slike består av bli kalt tverrsnitt. Tenk deg at en bunke papir utgjør et rektangulært prisme, så vil hvert stykke papir være et tverrsnitt av papiret. Ved å forestille oss hjernen bruker vi den samme tankegangen. MR (magnetisk resonansavbildning) (se informasjon om MR) fanger tverrsnittene i hjernen, og fra å bruke grensene definert i hvert "lag" i hjernen som tilbys, kan vi konstruere en struktur for å modellere og finne hjernens volum. Vi må imidlertid først bygge en funksjon for å gi informasjon om slike grenser.
Trinn 3: Konfigurere en funksjon: LevelCurveTracings.m
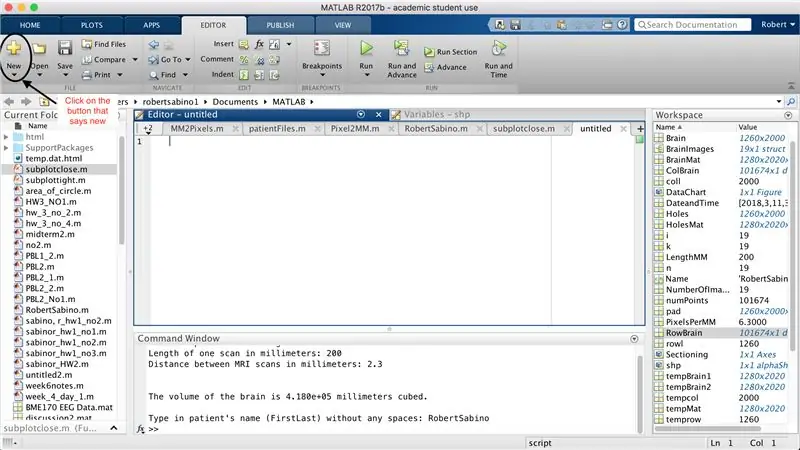
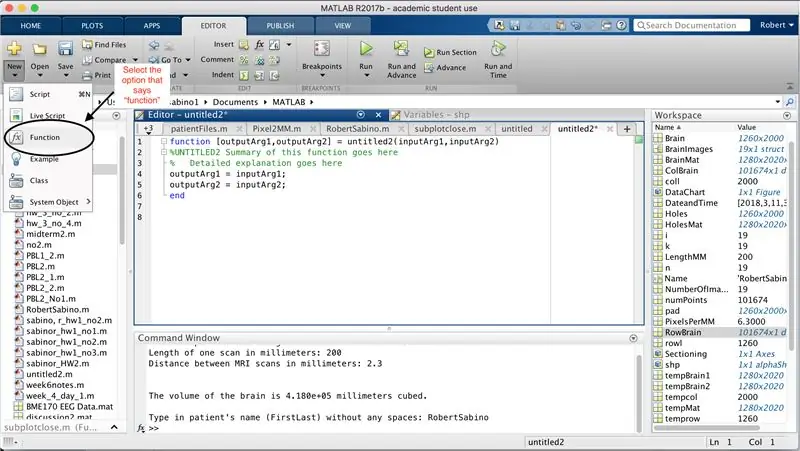
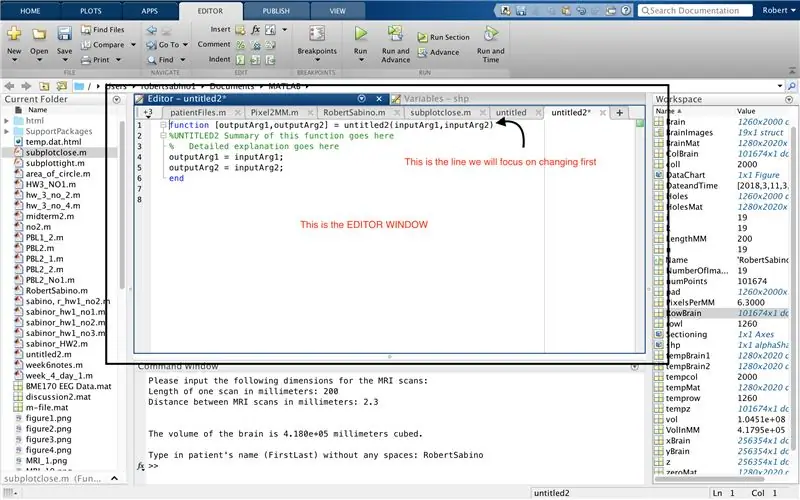

Først må du kontrollere at datamaskinen har MATLAB_R2017b lastet ned (last ned her) og åpne MATLAB. I MATLAB -grensesnittet klikker du på knappen i øvre venstre hjørne av vinduet som sier "nytt" med et sterkt gult pluss -tegn, og velger alternativet "funksjon" for å åpne et mellomrom i redigeringsvinduet som ligner det i tredje bildet. Vi vil fokusere på å endre den første linjen for å sette opp funksjonen. Der det står "outputArg1", erstatt det med "brain", "outputArg2" for å si "hull", "untitled2" til "exp2" og "inputArg1" til "image", og slett "inputArg2". Du har nå en funksjon som skal kalles ved å bruke "exp2", ta ett argument "image" og sette grensene for "hjerne" og "hull". Den første linjen i funksjonen skal ligne linjen som er avbildet i det fjerde bildet. Slett all koden under denne første linjen.
Trinn 4: Utvikle den bundne algoritmen: Finne grenser

Skriv inn koden som følger under linjen. Denne delen av funksjonen gjør følgende linje for linje.
- Last inn bildet "image" i variabelen "mri".
- Gjør "mri" til et bilde laget av verdier i en rekke tall til ener og nuller (aka binærisering) basert på en angitt terskelverdi. Hvis verdien i en piksel er lik eller større enn 0,1, er den satt til en, hvis ikke, er verdien på den pikslen satt til null.
- De følgende fire linjene gjør 10 kolonner og rader i kantene på MR -skannelaget til nuller, for å unngå å lese feil verdier som å gjøre grenser (som vi har lært ved å eksperimentere med koden).
- På den siste linjen sporer bwboundaries grensene for det binære bildet "mri" og setter det lik "b", en matrise med elementene hvis indekser tilsvarer grensene som er satt til ett.
Trinn 5: Utvikle den bundne algoritmen: Generere ytre bundet matrise
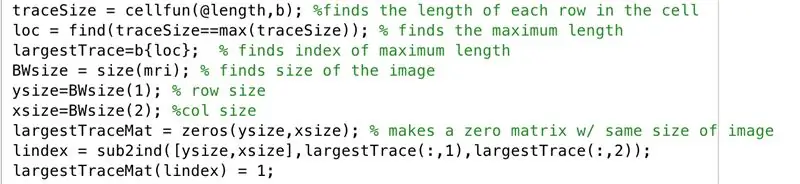
Følg med i redigeringsvinduet med følgende kode på bildet. Denne delen av koden gjør følgende linje for linje.
- Finn lengden på hver av radene i det binære bildet "b" (cellfun bruker funksjonslengden til hver rad).
- Sett "loc" for å lagre maksimal lengder.
- Finn indeksen for maksimal lengde, satt til å lagre i "LargestTrace".
- Finn størrelsen på bildet "mri", som består av samme størrelse som "b", og sett til "BWsize".
- Finn antall rader i bildens matrise, sett til "ysize".
- Finn antall kolonner i bildens matrise, satt til "xsize".
- Generer matrisen "grutsteTraceMat", en "ysize" av "xsize" matrise av nuller.
- Finn ekvivalentindeksen fra de abonnerte verdiene som tilsvarer der de størsteTrace x -verdiene og y -verdiene var, lagre i vektoren "lindex".
- I matrisen med nuller, "grutsteTraceMat", gjør du elementene ved indeksene som tilsvarer indeksverdier lagret som elementer i "lindex" til en.
Dermed har den logiske matrisen "LargestTraceMat" det største avgrensede området av det gitte hjerneskannertverrsnittet plottet som de med et bakteppe av nuller
Trinn 6: Utvikle den bundne algoritmen: Arbeide med senterpunkt

Deretter må vi teste for å se om tverrsnittet består av mer enn ett område (det største). Ved å teste justeringen av den største regionens sentroid, kan vi se om det er en sammenhengende region, som vil gi en mer sentrert sentroid, eller muligheten for flere regioner.
- Bruk "regionProps" for å finne informasjon om sentroidene som er tilstede, satt til strukturen "tempStruct"
- Form array "centroids" med data fra feltet "centroid" sammenkoblet vertikalt
- Ta den andre kolonneverdiene til "sentroider" (de horisontale dimensjonskoordinatene)
- Kjør et filter for å kontrollere justeringen av sentroiden til det horisontale senteret
Trinn 7: Utvikle den bundne algoritmen: Når en sentroid ikke er sentrert
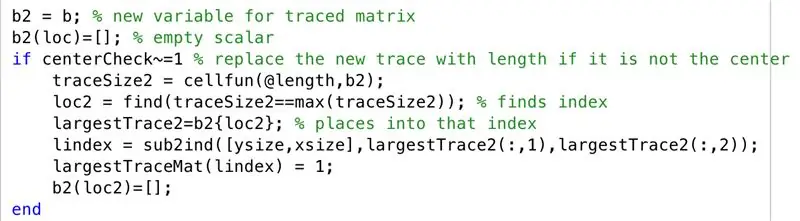
I scenariet om at centroiden til den største sporregionen ikke er sentrert, går vi gjennom følgende trinn. Som vi hadde observert i MR -skanningene, var tendensen å ha hjernehalvdeler avbildet i tverrsnittet når de ikke var sammenhengende, så vi fortsetter nå å plotte det nest største sporet sammen med det største sporet i "grutsteTraceMat"
- Sett den spores matrisen til en ny variabel "b2"
- Initialiser tom matrise "b2", med et sett indeksert av "loc"
- Lag en betinget, for når en sentroid ikke er sentrert (dvs. et lag med flere regioner)
- Angi en ny sporestørrelse for hver rad (traceSize2)
- Sett "loc2" for å finne indeksene der grenser er tilstede
- La celler spesifisert med "loc2" i "b2" være lik "grutsteTrace2"
- Konverter abonnementene til indekser, satt til "lindex"
- Endre elementer som tilsvarer "lindex" i "LargestTraceMat" til 1
- Initialiser tom matrise "b2", med et sett indeksert av "loc2"
Trinn 8: Utvikle den bundne algoritmen: Interferens av hull

Ved håndtering av hull holdt verdiene lagret i "b2" oversikt over andre strukturer enn det største sporet, og plottingen av disse på en fylt form av "grutsteTraceMat" vil avsløre hvor det er hull i hjernegruppene.
- Lag matrise "filledMat", som er en fylt i form av "LargestTraceMat"
- Lag matrisen "interferenceMat", en "ysize" av "xsize" matrisen med nuller
- Lag matrise "interferenceloc", for å lagre verdiene fra "b2", sammenkoblet vertikalt
- Lag array "lindex" for å lagre indeksene som tilsvarer "interferenceloc"
- For indekser i "interferenceMat" som tilsvarer "lindex", sett verdien til 1, noe som gjør et annet avgrenset område
Trinn 9: Utvikle den bundne algoritmen: Finne hull, fullføre hjernegrenser og hullgrenser

- Sett matrisen "tempMat" lik "interferenceMat" pluss "filledMat", og legg dermed hver verdi i matrisen til hverandre
- Sett matrisen "holesLoc" lik indeksene der "interferenceMat" og "filledMat" begge var lik en
- Sett opp "holesMat" som en nullmatrise med dimensjonene "ysize" x "xsize"
- Sett indekser i "holesMat" som er lik "holesLoc" som de
- Sett "hjerne" til "grutsteTraceMat"
- Sett "hull" til "hullmat"
Med funnet hvor verdiene til de tilsatte matrisene var lik 2, ble hullplasseringene lett sikret og plottet på en tom matrise.
Trinn 10: Loggdata: Funksjon PatientFiles.m
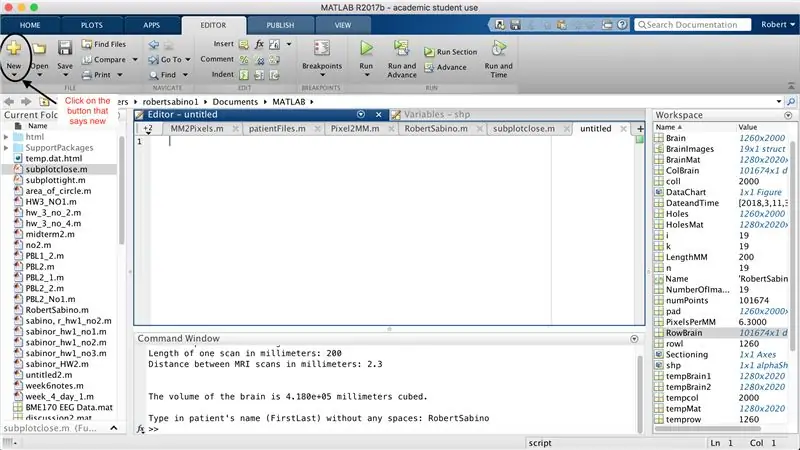
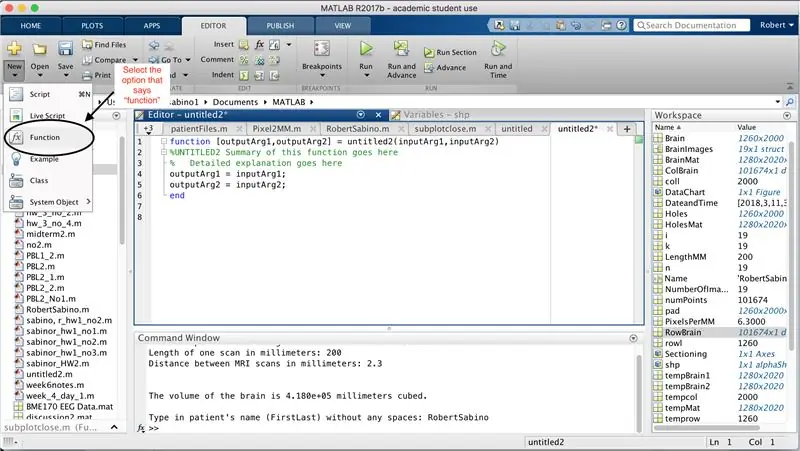
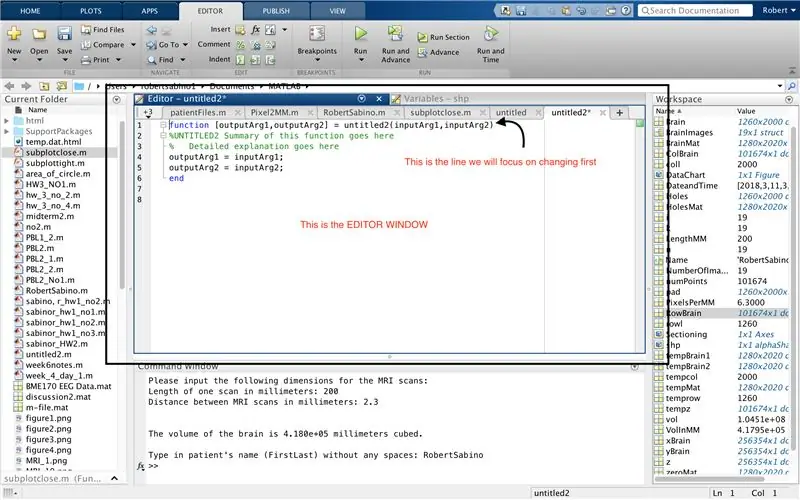

I likhet med oppsettet av den siste funksjonen, klikker du på knappen i øvre venstre hjørne av vinduet som sier "Ny" med et sterkt gult pluss -tegn, og velger alternativet "funksjon" for å åpne et mellomrom i redigeringsvinduet ligner det på det tredje bildet. På den første linjen, slett utmatrisen og erstatt med bare "output", erstatt "untitled2" med "patientFiles", slett alle inputargumentene, og følg i stedet formateringen som er angitt i det fjerde bildet av kodelinjen. Den første linjen i denne funksjonen skal matche formateringen av bildet.
Trinn 11: Logge data i filer
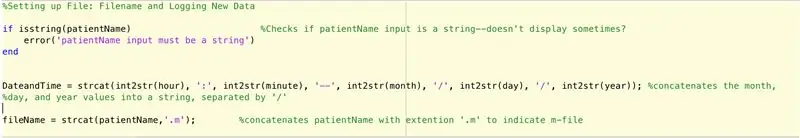

For å sette opp en fil for å logge dataene som er funnet av hovedfunksjonen (som ikke skal beskrives), må vi følge disse trinnene (som foreskrevet av koden linje for linje).
- Sjekk om input for patientName er en streng.
- Hvis det ikke er en streng, må du vise at pasientnavn -input skal være en streng.
- Avslutt if -setningen (forhindre feil).
- Sett opp en strengsetning "DateandTime" som vil gi følgende format: time: minutter-måned/dag/år.
- Sett variabel filnavn til følgende: pasientnavn.m.
Nå til neste del av funksjonen: Finnes det allerede en fil med dette navnet?
1) Anta at filen med dette navnet allerede eksisterer:
- Kjør filen for å få verdiene fra tidligere i kø
- Legg til "DateandTime" -dataene for den gjeldende iterasjonen som en ny celle i cellearrayen med x -verdier (indeksenden+1)
- Legg til den nåværende "brainVolume" -verdien som en ny celle i cellearrayet med y -verdier (indeksenden+1)
- Lagre de nåværende variablene som er lastet inn i filen.
2) Anta at filen med dette navnet ikke eksisterer:
- Lag en ny fil med navnet lagret i variabelen "pasientnavn"
- Legg til gjeldende "DateandTime" -data som en celle i det tomme cellearrayet med x -verdier
- Legg til gjeldende "brainVolume" -data som en celle i det tomme cellearrayet med y -verdier
- Lagre de nåværende variablene som er lastet inn i filen.
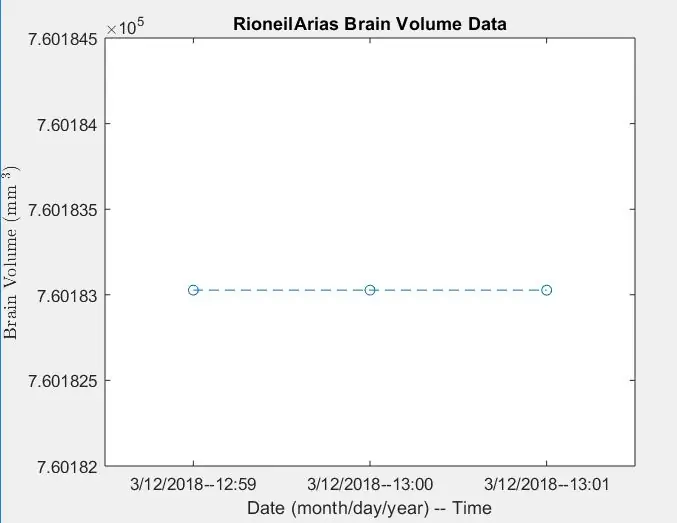
Trinn 12: Loggdata: Visning av en plott av hjernevolum over tid

- Konverter x -verdier -arrayet (xVals) til et kategorisk array (xValsCategorical), for å tillate plotting
- Generer figurvindu 5
- Plott punktene angitt med "xValsCategorical" og "yVals" (som inneholder hjernevolum), ved hjelp av hule sirkler for å indikere punkter og for å være forbundet med stiplete linjer
- Tittel plottet som: patientName Brain Volume Data
- Merk x -aksen som vist på bildet
- Merk y -aksen som vist på bildet
- La figur 5 være lik utgang
Fra dette vil funksjonen pasientnavn som blir kalt, gi en fil med redigerte data som holder styr på hjernevolum over tid, og et plott som viser trender.
Trinn 13: Lukking av hull i delplott: Subplotclose.m

Funksjonen, tilpasset fra kode fra https://www.briandalessandro.com, fungerer for å lukke hullene mellom delplottfigurene til hovedkoden, når figurene som viser MR -bildene og hjernelagene opprettes. Subplot -funksjonen som brukes i subplotclose.m justerer posisjonen til de gitte subplotene slik at de passer godt til hverandre i aspektet av den lengre dimensjonen. For eksempel, hvis koden har til hensikt en 7 x 3 -matrise, vil radene passe godt ettersom raddimensjonen er lengre. Hvis koden har til hensikt en 3 x 7 matrise, vil kolonnene passe godt, med hull i radene, som vist i figurene i vår hovedkode.
Trinn 14: Hovedkoden: Fjern alle og be om innspill

For å starte hovedkoden, klikk på den samme knappen som sier "Ny" i øvre venstre hjørne av vinduet, og velg "Skript" i stedet for "Funksjon" fra de tidligere seksjonene. Skriv inn koden som vist på bildet i redigeringsvinduet. Kodelinjene gjør følgende oppgaver i rekkefølge:
- Lukk alle åpne filer unntatt 0, 1 og 2.
- Lukk alle figurvinduer.
- Fjern alle variabler i arbeidsområdet.
- Fjern kommandovinduet.
- Vis i kommandovinduet: Skriv inn følgende dimensjoner for MR -skanningene:
- På en ny linje i kommandovinduet, spør: Lengde på en skanning i milimeter:. Svaret som legges inn av brukeren vil bli satt til variabelen "lengthMM".
- På en ny linje, spør: Avstand mellom MR -skanninger i millimeter:. Svaret som legges inn av brukeren vil bli satt til variabelen "ZStacks".
Trinn 15: Hovedkoden: Batchbehandling av bildene

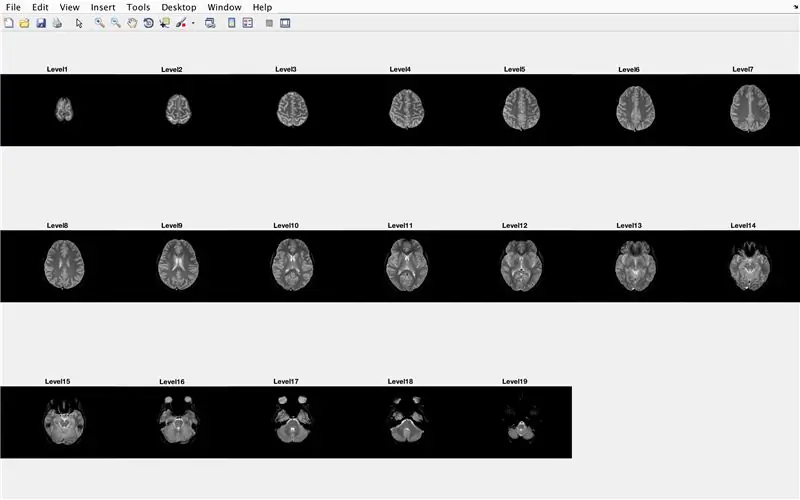
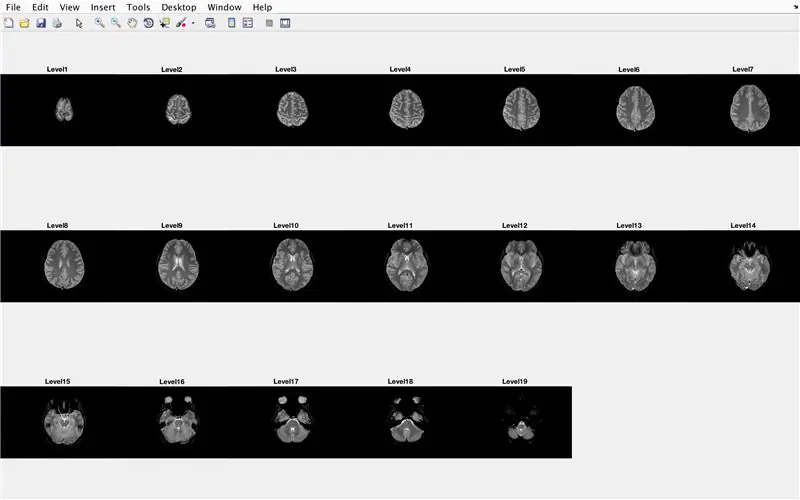
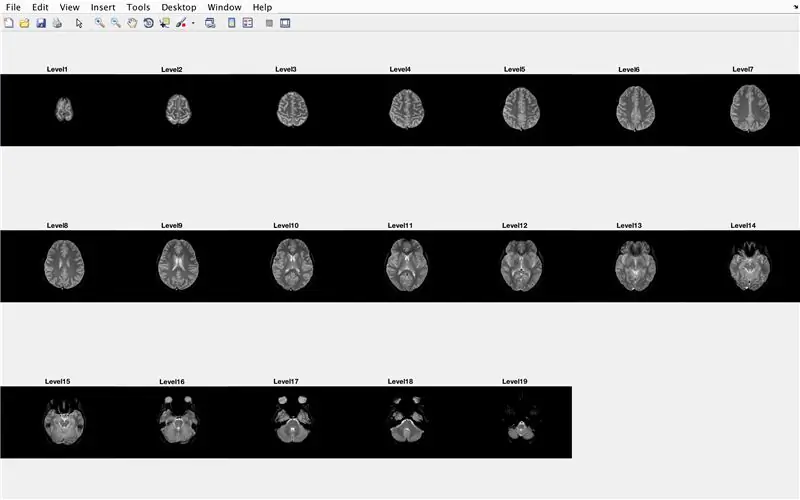
I denne delen vil koden laste opp bildene (bestående av MR -skanninger av tverrsnittene i hjernen) og lagre navnene på hver bildefil i variabelen "Base" og vise hver av MR -skanningene. Følg med koden på bildet, som gjør følgende:
- Lag strukturmatrise "BrainImages" som inneholder informasjon om alle filene i den nåværende mappen som passer til navneformatet til MRI _ (). Png
- Sett variabel "NumberofImages" lik antall elementer i strukturmatrisen "BrainImages"
- Åpne figurvindu 1
- Sett en for -løkke for å bla gjennom for antall bilder som telles i filen
- For hver sløyfe er "CurrentImage" det respektive navnet på hver fil MRI_i.png, med iterasjonsnummeret som 'i'
- Lag en tomt på 3 x 7 for å vise de 19 bildene som skal lastes inn av "imshow"
- Vis hvert bilde som et annet element i delplottfigurvinduet
- Tittel hvert delplot -element som Nivå_, der tomt er iterasjonstallet for for -løkken.
- Slutt for -løkken (unngå feil)
Dette vil vise i figurvindu 1 alle MR -skanningene i rå form i en 3 x 7 -konfigurasjon uten hull i x -retningen.
Trinn 16: Hovedkoden: Polstring

Med polstring unngår vi problemet med små avvik i bildestørrelsene som kan gi feil ved dimensjonsforskjell i tilfelle at ett bilde er litt større enn et annet.
- Åpne figurvindu 2
- Last inn bildematrisen fra MRI_1-p.webp" />
- Finn størrelsen på bildets matrise og sett til "OriginalXPixels" (for antall rader) og "OriginalYPixels" (for antall kolonner)
- Sett opp matrisen "BrainMat" for å bestå av alle nuller med 20 flere rader og 20 flere kolonner for hvert plan, og 19 totale tverrsnitt, en per plan.
- Sett opp "HolesMat" for å bestå av den samme tredimensjonale gruppen med nuller for å legge inn hullkoordinater senere
- Lag "zeroMat" til størrelsen på puten pluss tjue rader og tjue kolonner, et todimensjonalt utvalg av nuller.
Trinn 17: Hovedkoden: Bestemmelse av grenser
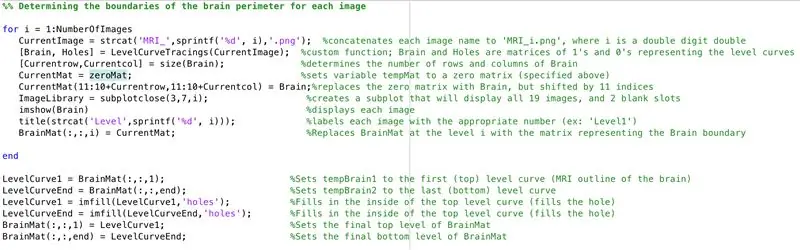
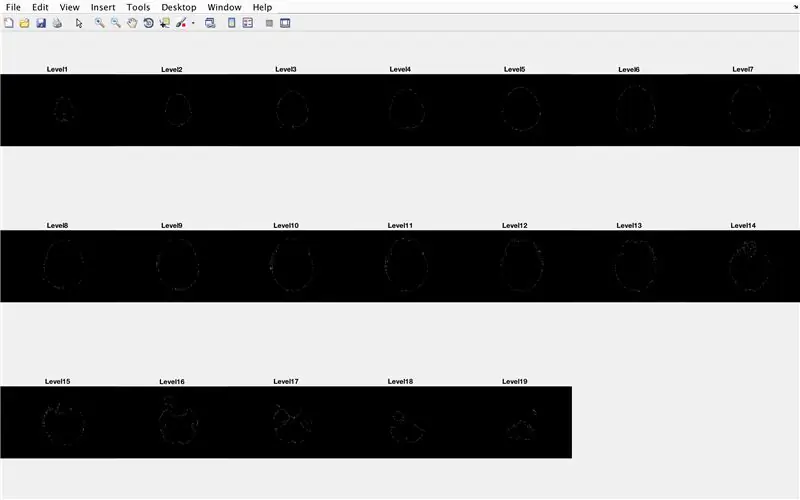
- Sett en for loop for å gå gjennom dataene fra hvert bilde som er lastet inn tidligere
- På samme måte som batchbehandling tidligere, laster "CurrentImage" opp filer med "MRI_i.png", hvor i er iterasjonstallet
- Kjør hvert bilde gjennom behandlingsfunksjonen "LevelCurveTracings2.m" som du har laget tidligere
- Finn størrelsen på utgangen "Brain", sett antall rader til "Currentrow" og antall kolonner til "Currentcolumns"
- Sett "CurrentMat" til en matrise med nuller med dimensjonene angitt av "Currentrow" og "Currentcolumns"
- Sentrer dataene fra "Brain" i "CurrentMat", med en margin på 10 rader på alle sider
- Lag en delplott med dimensjoner 3 x 7, for å vise grensene for bildene
- Tittel på hvert av delplotelementene i figurvinduet
- Generer tredimensjonal matrise "BrainMat" som består av hvert grenselag "CurrentMat"
- Avslutt for -løkken (for å unngå feil)
Delen nedenfor fyller hullene igjen øverst og nederst i den foreslåtte tredimensjonale formen
- Sett "LevelCurve1" lik det første laget av "BrainMat" (bunnen av solid)
- Sett "LevelCurveEnd" lik det siste laget av "BrainMat" (toppen av solid)
- Overskrive "LevelCurve1" med et fylt lag
- Overskrive "LevelCurveEnd" med et fylt lag
- Sett det utfylte laget som det nederste laget av "BrainMat"
- Sett det utfylte laget som det øverste laget av "BrainMat"
Trinn 18: Hovedkoden: Bestemmelse av riktig Z -dimensjon

De tre første linjene består av å sette opp et tomt array "z", og gjøre enkle konverteringsoperasjoner (dele piksler med lengde) for å få en riktig avlesning av volum i mm^3
- Lag en for -løkke for å bla gjennom hvert lag
- Finn antall en i et gitt lag
- Konverter z -koordinatene for de til verdier som skaleres til riktig forhold, satt til "tempz", en kolonnevektor
- Legg til z -verdi for nivåkurven til vektoren z
Med dette er z -koordinatene justert riktig.
Trinn 19: Hovedkoden: Bestemmelse av X- og Y -koordinater

Nå for å bestemme x- og y -posisjonene til hvert av punktene i grensene.
- Initialiser "xBrain" som en tom matrise
- Initialiser "yBrain" som en tom matrise
- Sett opp en for loop for å gå gjennom hvert bilde som er lastet inn
- Kompiler en to -kolonne matrise for å lagre de plane koordinatene til hvert punkt i grensen, representert med kolonnevektorene "RowBrain" og "ColumnBrain"
- Legg til "xBrain" med de nåværende "RowBrain" -koordinatene
- Legg til "yBrain" med de nåværende "ColumnBrain" -koordinatene
- Avslutt for -løkken (for å unngå feil)
Trinn 20: Hovedkoden: Plotte en tredimensjonal struktur, finne volum og loggdata
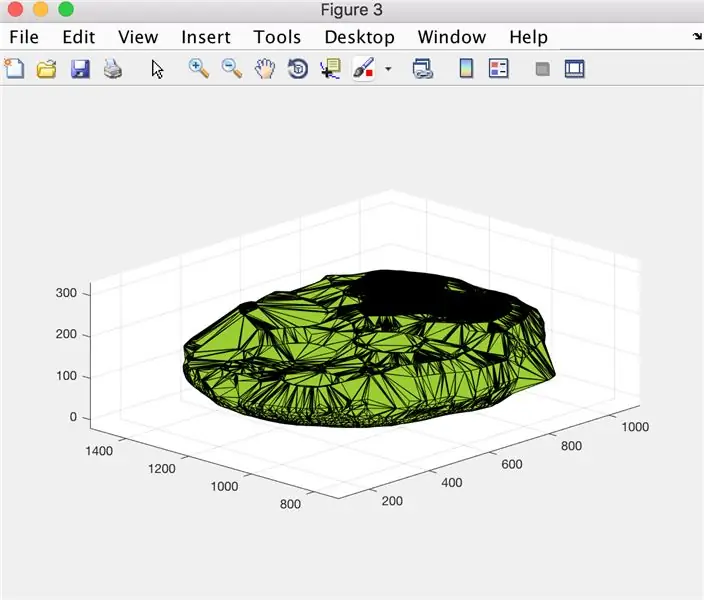

Ved å bruke funksjonen alphaShape, vil vi lage en tredimensjonal struktur som vi kan beregne volumet av hjernen fra.
- Bruk funksjonen alphaShape, plugg inn vektorene "xBrain", "yBrain" og "z" for x-, y- og z -koordinatene, og sett lik "BrainPolyhedron"
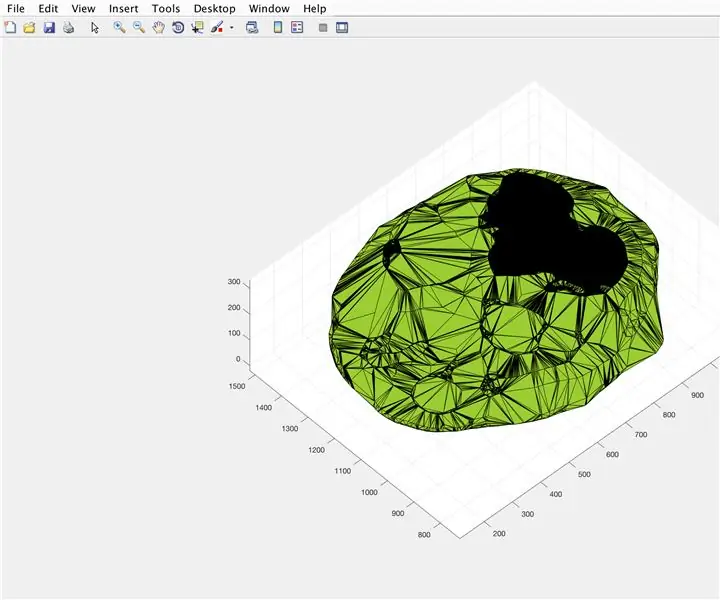
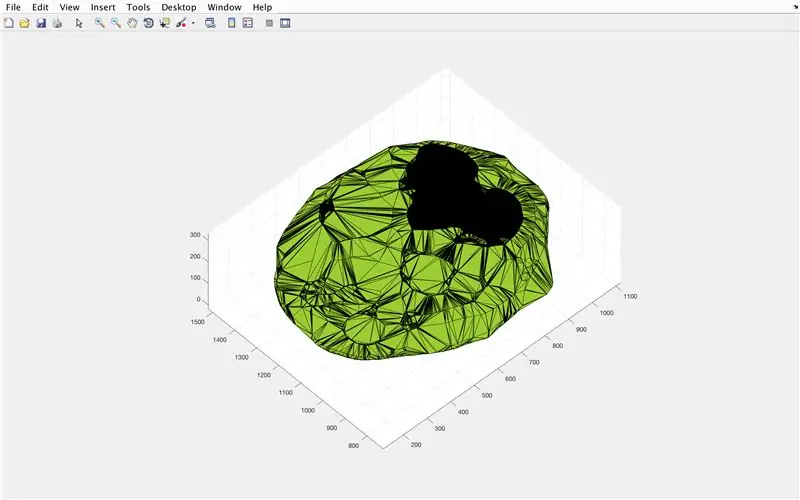
- Åpne figurvindu 3
- Plott den beregnede alfa -formen "BrainPolyhedron", som vises i figurvinduet
- Beregn volumet til alfa -formen ved å bruke en funksjon "volum" som fungerer for alfa -former
- Konverter volumet til mm^3
- Skriv ut volumet på det faste stoffet i kommandovinduet
- Be om at et pasientnavn skal angis som inndata
- Få gjeldende dato og klokkeslett med klokken, og sett til "DateandTime"
- Ring funksjonen "patientFiles" for å logge og plotte dataene som er beregnet
Herfra skal det andre og tredje bildet vise figurene som dukker opp, og det fjerde bildet det som skal vises i kommandovinduet.
Anbefalt:
ATtiny85 Wearable Vibrating Activity Tracking Watch & Programming ATtiny85 With Arduino Uno: 4 Steps (with Pictures)

ATtiny85 Wearable Vibrating Activity Tracking Watch & Programming ATtiny85 With Arduino Uno: How to make the wearable activity tracking watch? Dette er en bærbar gadget designet for å vibrere når den oppdager stagnasjon. Bruker du mesteparten av tiden din på datamaskinen som meg? Sitter du i timevis uten å innse det? Da er denne enheten f
Time Cube - Arduino Time Tracking Gadget: 6 trinn

Time Cube - Arduino Time Tracking Gadget: Jeg vil foreslå deg et enkelt, men veldig nyttig arduino -prosjekt for å spore tidshendelser ved å snu en smart kube -gadget. Vend den til " Arbeid " > " Lær " > " Husarbeid " > " Hvile " siden, og det vil telle
IOT: ESP 8266 Nodemcu Controlling Neopixel Ws2812 LED Strip Over Internet Using BLYNK App: 5 Steps

IOT: ESP 8266 Nodemcu kontrollerer Neopixel Ws2812 LED -stripe over internett ved hjelp av BLYNK -appen: Hei folkens, i denne instruksjonene laget jeg et lys ved hjelp av neopixel led -stripe som kan kontrolleres over internett fra hele verden ved hjelp av BLYNK -appen og nodemcu er fungerer som hjernen i dette prosjektet, så lag ditt omgivende lys for deg
Er det en hånd? (Raspberry Pi Camera + Neural Network) Del 1/2: 16 trinn (med bilder)

Er det en hånd? (Raspberry Pi Camera + Neural Network) Del 1/2: For noen dager siden skadet jeg høyre håndledd på treningsstudioet. Etterpå hver gang jeg brukte datamusen, forårsaket det mye smerte på grunn av bratt håndledningsvinkel. Det var da det slo meg " ville det ikke vært flott hvis vi kunne konvertere en hvilken som helst overflate til en trackp
Arduino Neural Network Robot: 21 trinn (med bilder)

Arduino Neural Network Robot: Denne instruksjonsbasen er basert på en 3 -delt serie jeg laget for Make YouTube Channel som viser deg nøyaktig hvordan du prototype, designe, montere og programmere din egen Arduino neurale nettverksrobot. Etter å ha sett hele serien, bør du ha en innsats
